Comparação de métodos para
O isolamento de ácidos nucléicos de sementes intactas requer o rompimento mecânico da semente seguido pela extração e subsequente purificação do ácido nucléico. A ruptura mecânica é frequentemente realizada manualmente com um almofariz e pilão, uma abordagem que não é prática para a triagem de alto rendimento de sementes, pois a moagem manual é lenta e a reutilização de almofariz e pilões pode levar à contaminação cruzada. Alternativamente, os ácidos nucleicos podem ser isolados de sementes em um formato de placa de micropoços usando um moinho de bolas que interrompe mecanicamente as sementes.
As metodologias de isolamento convencionais podem então ser usadas para purificar os ácidos nucleicos dos homogenatos de sementes. A eficiência da quebra de sementes depende do tipo de moinho de bolas usado no processo de moagem. Os moinhos de contas padrão adaptados a placas de micropoços são modelados a partir de “agitadores de tinta” e movem as placas em um movimento de “oito”. Este movimento não leva à ruptura uniforme da semente. O Geno / Grinder® é projetado para romper com eficácia os materiais celulares, oscilando a placa verticalmente. Este movimento permite que as bolas impactem as sementes mais diretamente do que os moinhos convencionais, onde as bolas impactam as paredes do poço além da semente.


A soja que é embebida durante a noite em água é homogeneizada de forma eficaz e uniforme em menos de 3 minutos. A polpa resultante pode então ser usada como fonte de DNA para análise genética.
Materiais e
Após homogeneização por 40 seg. usando o lisador de células competitivo, DNA de peso molecular inferior (qualidade inferior) foi obtido para amostras processadas em qualquer um dos quatro tempos de execução usando o Geno / Grinder. Aos 90 e 120 segundos, quase todo o DNA isolado usando o Geno / Grinder era superior a 6.557 pb, indicando que cisalhamento ou degradação significativa não ocorreu, mesmo no tempo de processamento mais longo. Pouca ou nenhuma diferença nos resultados foi observada nesses dois tempos de processamento. Enquanto os tempos de execução são de 40 e 60 seg. no Geno / Grinder produziu DNA de alto peso molecular, quantidades insuficientes foram obtidas.


Purificação de DNA usando o
Após a trituração, a soja foi misturada com 400 µL de tampão AP1 e 4 µL de solução estoque de RNase. A mistura foi incubada durante 10 minutos a 65° C. O tampão AP2 (130 µL) foi então adicionado ao lisado, misturado e incubado durante 5 minutos em gelo. Os precipitados foram removidos com a coluna de rotação QIAshredder ™. O eluente foi misturado com 0,5 volume de tampão AP3 e 1 volume de etanol (96 -100%). Após a mistura, o lisado foi aplicado à mini coluna spin DNeasy e centrifugado a 9000 rpm por 1 minuto. Durante esta etapa, o DNA se liga à coluna. O fluxo é descartado. A coluna foi lavada com tampão AW e depois centrifugada à velocidade máxima para secar a membrana. O DNA foi eluído com 50 µL de Tampão AE incubando por 5 minutos em temperatura ambiente e depois centrifugando por 1 minuto a 9000 rpm.
Kit de
Após a trituração, a soja foi misturada com 600 µL de solução de lise de núcleo e incubada por 15 minutos a 65° C. RNase foi adicionado seguido por uma incubação de 15 minutos a 37° C e um resfriamento de 5 minutos até a temperatura ambiente. A solução de precipitação de proteína foi adicionada seguida de centrifugação para precipitar as proteínas do pellet. O sobrenadante contendo o DNA foi transferido para outro tubo de microcentrífuga contendo isopropanol. A amostra foi misturada suavemente por inversão e depois centrifugada a 9000 rpm. O sobrenadante foi removido e o sedimento foi lavado com etanol a 70%. O tubo foi centrifugado e o etanol foi decantado. O excesso de etanol foi removido invertendo o tubo sobre papel absorvente e secando ao ar durante 15 minutos. O DNA foi ressuspenso em 50 µL de Solução de Reidratação de DNA durante a incubação por 1 hora a 65° C.


Método CTAB para
A soja moída foi diluída em 500 µL de tampão CTAB (20 g CTAB / L, NaCl 1,4 M, Tris / HCl 0,1 M, EDTA 20 mM). Esta mistura foi incubada durante 30 minutos a 65° C. Foi então centrifugada durante 10 minutos a 9000 rpm. A camada superior foi extraída com igual volume de clorofórmio. Após mistura durante 30 segundos, a mistura foi centrifugada durante 10 minutos a 9000 rpm. O sobrenadante foi transferido para um novo tubo, e dois volumes de solução de precipitação CTAB (5 g CTAB / L, 0,04 M NaCl) foram adicionados. A mistura foi incubada durante 60 minutos à temperatura ambiente e depois centrifugada durante 5 minutos a 9000 rpm. O sobrenadante foi removido e o precipitado foi dissolvido em 1,2 M. NaCl. Esta solução foi extraída com clorofórmio. A camada superior (fase aquosa) foi transferida para um novo tubo e 0,7 volumes de isopropanol foram adicionados. A solução foi então congelada durante 20 minutos a -80°C. A amostra foi descongelada à temperatura ambiente e centrifugada. O pellet de DNA resultante foi lavado com etanol 70% gelado e centrifugado. O etanol foi descartado e o etanol residual foi removido com um DNA SpeedVac. O pellet de DNA foi ressuspenso em tampão TE.
Purificação de DNA usando
Uma comparação dos rendimentos de DNA para os procedimentos de isolamento foi feita por eletroforese em gel de agarose (0,7% de agarose em tampão TAE 1X) que é ilustrada na Figura 1.
O DNA genômico foi testado para sequências geneticamente modificadas, ou seja, análise de OGM, por meio de PCR. As amostras foram analisadas para o gene da lecitina de soja e o promotor 35S do vírus do mosaico da couve-flor / marcador de sequência de transcrição de Petúnia encontrados em soja geneticamente modificada (Roundup ReadySoya). A análise das reações foi feita eletroforeticamente (1X TAE com 3% de agarose) e é ilustrada na Figura 2.

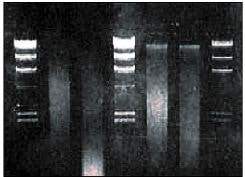
Figura 1. Comparação de métodos para isolamento de DNA de soja. Pistas 1, 4 e 7 – digestão de Lambda Hindill, Pista 2 – Kit Qiagen Genomic DNA, Pista 3 – Método CTAB, Pista 5 – Kit Promgea Wizard, Pista 6 – Kit Qiagen DNeasy.

Figura 2. Análise de PCR de DNA isolado de soja
Resultados e
O método de isolamento usado para extrair DNA do homogenato de soja afeta a quantidade e a qualidade do DNA genômico isolado. A eletroforese em gel de agarose do DNA genômico (fig. 2) ilustra uma diferença significativa no rendimento e no tamanho do fragmento do DNA. Com base na fluorescência do esfregaço de DNA no gel corado com brometo de etídio, o método CTAB produziu o maior rendimento, mas o tamanho do fragmento é extremamente pequeno em comparação com as outras preparações de DNA e medido em relação aos marcadores de peso molecular. Dependendo da aplicação do DNA, rendimentos mais baixos de fragmentos de grande porte (por exemplo, o método Wizard ou o método da coluna genômica) podem ser preferidos.
A análise de PCR das amostras foi positiva para o gene da lectina de soja e negativa para o promotor 35 S / sequência TS de Petúnia. Esta análise demonstra que homogenatos de soja gerados a partir do Geno / Grinder são adequados para análise genética. Isso é particularmente útil para análises PCR de alto rendimento em laboratórios de biotecnologia agrícola ou laboratórios que realizam análises de OGM.
Tire suas dúvidas ou
Solicite sua cotação aqui






